The Mass Spectrometry Platform (MaSUN) at the University of Namur specializes in mass spectrometry analyses, covering proteomics and the identification of small molecules.
It supports academic and industrial projects, from sample preparation to data analysis, with recognized expertise in quantification, molecular characterization, and metabolomic studies.
Presentation of the MaSUN platform
MaSUN brings together equipment from several research units at the University of Namur:

Areas of expertise
- Comprehensive proteomic analyses: extraction, digestion, nanoLC-MS/MS, DDA and DIA, data processing
- Identification and characterization of organic compounds and biomolecules
- Studies of post-translational modifications (PTM) and comparative analyses
News
- Launch of metabolomics with Professor Marc Hennequart
- 2024: Upgrade from timsTOF Pro to timsTOF HT and from nanoElute to nanoElute 2 thanks to funding from UNamur and with the help of URBC.
- 2018: Acquisition of a high-resolution ion mobility spectrometer for quantitative proteomics thanks to a grant from the FNRS.
Key equipment
The MaSUN platform combines various complementary mass spectrometers:
- Waters ACQUITY QDa with a Waters Alliance HPLC for routine chemical analysis.
- Bruker MaXis Impact coupled with an HLPC, Thermo Ultimate 3000 2D-LC mainly dedicated to metabolomics, proteomic profiling, and high-resolution analysis of small molecules.
- Bruker timsTOF coupled with a Bruker nanoElute dedicated to quantitative proteomic analyses.
Available software
The following software programs, designed for proteomic data processing, protein identification, and quantification, are available to meet various sample analysis needs:
- DIA-NN, DIA-Analyst (Monash University)
- MaxQuant, Perseus
- Mascot Server 2.8
- PEAKS 13 (LFQ, SILAC, PTM, De Novo sequencing)
- Scaffold 5 (Spectral Counting)
- Merox 2.0 (Cross-linking)
Services and collaboration
The platform offers:
- Training and support for researchers: protein extraction, electrophoresis where applicable, enzymatic digestion, data analysis
- Comprehensive analyses for academic projects
- Services for companies with integrated services

We explain it to you in pictures

Spotlight
News
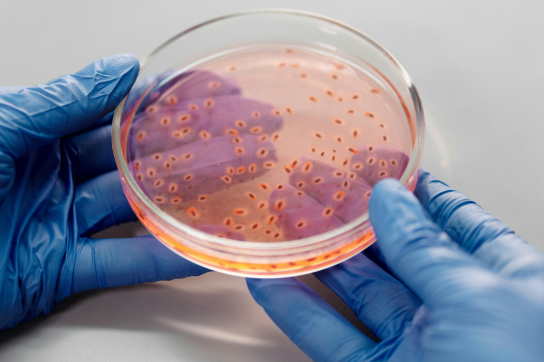
A prestigious publication for an international microbiology research team
A prestigious publication for an international microbiology research team
The team of professor Xavier De Bolle has just published an article in the prestigious EMBO Journal published by Springer Nature. His discovery? A lipid transport channel through the cell membrane of Brucella, the bacteria responsible for Brucellosis in cattle. This finding could be used to generate attenuated strains of the bacteria; a process used in vaccine manufacturing.
The study of Brucella is one of the areas of microbiology in which UNamur research teams have specialised for more than twenty years. This pathogenic bacteria infects livestock, causing Brucellosis, a disease that can be transmitted to humans through the consumption of unpasteurised dairy products, contact with infected animal tissue or inhalation. It is one of the most widespread zoonoses in the world.
A catalogue of weaknesses
Over the years of research, the team led by Professor Xavier De Bolle, Professor in the Department of Biology, member of the Microorganism Biology Research Unit (URBM) at the University of Namur and investigator at the WEL Research Institute, has been able to establish a kind of “catalogue” of Brucella's weaknesses.
There are millions of species of bacteria. It is therefore impossible to study them on a case-by-case basis. However, they have similar behaviours and functions, which is why a specific bacterium is considered the model: Escherichia coli (or E. coli), an intestinal bacteria found in warm-blooded organisms. Discovered in 1885, it has been the subject of numerous studies and has therefore become a reference in scientific literature.
A bacterium such as E. coli or Brucella is like an onion with only two layers (inner and outer membrane) and a central element (the cytoplasm) in which the organism produces everything it needs. It also exports elements to the outside, particularly lipids. However, these processes are still poorly understood, even for E. coli. Nevertheless, they represent an interesting avenue to explore to reach the heart of the pathogenic bacteria.
Mutants pave the way
During this research, which combined bacteriology and genetics, the researchers developed a technology that allows them to induce mutations in Brucella and also to identify where these mutations are located. They succeeded in creating a mutant that showed them the way to a veritable ‘lipid tunnel’, a passageway in the inner and outer membrane walls of the bacteria.
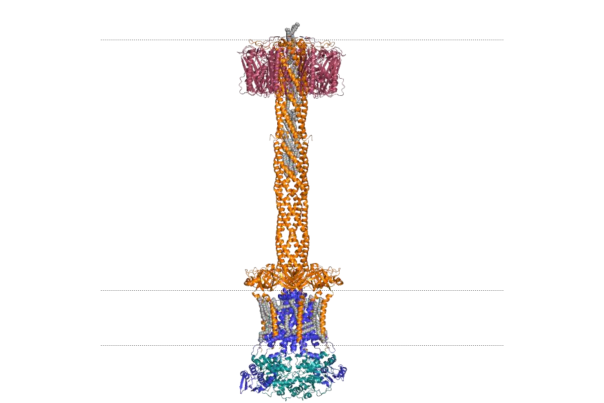
This provides valuable information about a new weakness in the pathogen and therefore provides information on how to combat it more effectively.

Our research, funded by the FNRS and the WEL Research Institute, is fundamental. Of course, we are still in the very early stages of developing applied processes. But we are taking another step towards unlocking the secrets of Brucella's armour so that one day we can hope to control the disease with an effective vaccine
An international research team
- Adélie Lannoy (currently post-doc in Toulouse) and Xavier De Bolle (URBM, UNamur – Belgium)
- Marc Dieu and Patsy Renard (UNamur, URBC - Belgium)
- Antonia García Fernández (Centro de Metabolómica y Bioanálisis (CEMBIO), Facultad de Farmacia, Universidad San Pablo-CEU – Spain)
- Raquel Condez-Alvarez (Department of Microbiology and Parasitology, Instituto de Investigación Sanitaria de Navarra (IdiSNA)University of Navarra – Spain)
The researchers would like to thank the FNRS and the WEL Research Institute for their support in this research. They would also like to thank the ‘Mass Spectrometry – MaSUN’ platform and the ‘Level 3 Biosafety Laboratory – BL3’ technology platform at UNamur for their cutting-edge equipment and expertise.
Xavier De Bolle - Mini CV
Xavier De Bolle presented his doctorate at UNamur (FUNDP at the time) in 1995. After 4 years of post-doc, including one at the University of Oxford, he joined URBM (Biology, Sciences), where he leads a research group. He has supervised 25 doctoral theses (including 4 in progress) and some of his former students have generated their own research team (at the UNamur, the UCLouvain, the VUB).
Professor Xavier De Bolle is a member of the URBM in the Department of Biology at the Faculty of Science of the University of Namur. He is a member of the Namur Research Institute for Life Sciences (Narilis).
Since June 2024, he is also a researcher at the WEL Research Institute for the Welbio-Health programme in the field of ‘Microbiology and Infectious Diseases’.
This work illustrates the cutting-edge expertise developed at UNamur in the field of molecular microbiology, an area in which UNamur has decided to invest by organising a master's degree in molecular microbiology since September 2019. This is a unique master's degree in Europe, taught entirely in English and rooted in research.
Find out more
- New clues to break through Brucella's armour (02/27/2023) - https://unamur.be/fr/newsroom/de-nouveaux-indices-pour-percer-larmure-de-brucella
- Alison Forrester and Xavier De Bolle awarded grants from the latest FRFS-Welbio call for projects (07/23/2024) - https://unamur.be/fr/newsroom/laureats-dernier-appel-projets-frfs-welbio

Parchment bindings under the microscope
Parchment bindings under the microscope
To restore an old book correctly, it is essential to know the secrets of its manufacture and the reasons for its deterioration. Thanks to the King Baudouin Foundation's Jean-Jacques Comhaire Fund, the restoration workshop of the Moretus Plantin University Library has launched a new research project on parchment bindings in the Southern Netherlands in the 16th and 17th centuries. The aim is to gain a better understanding in order to improve conservation.

La Bibliothèque Universitaire Moretus Plantin (BUMP) a la chance de bénéficier de son propre atelier de restauration, qui permet à la fois une conservation optimale des ouvrages et leur mise à disposition des scientifiques et du public dans de bonnes conditions. « En restauration, on doit généralement constater les détériorations sans connaître leurs origines. Pourtant, comprendre le mouvement séculaire d’une reliure permettrait d’adapter nos interventions afin qu’elles soient les plus adéquates vis-à-vis de cette évolution », explique Catherine Charles, restauratrice responsable de l’atelier de la BUMP.
Le nouveau projet financé par le Fonds Jean-Jacques Comhaire, qui soutient la recherche dans le domaine de l’archéométrie, vise à apporter des réponses aux questions que se posent les restaurateurs sur les reliures souples en parchemin réalisées dans nos régions aux Temps Modernes. « La BUMP conserve un corpus représentatif de ces reliures, et en particulier de reliures couvrant des volumes qui ont en commun le fait d’avoir été imprimés à Anvers, chez Plantin-Moretus, aux 16e et 17e siècles », indique Catherine Charles. « Dans cet ensemble cohérent de plus de 200 livres, nombreux sont les cas où une dégradation similaire est constatée : une courbure du dos de plus en plus accusée, ce qui fend les mors et fait ressortir le corps d’ouvrage. Le projet de recherche est l’opportunité de comprendre la construction matérielle de ces livres et de faire le lien ou non avec leur dégradation progressive ».
Le Fonds Comhaire rend possible l’engagement, aux côtés de Catherine Charles, d’Ana Oñate Muños, assistante de recherche diplômée de La Cambre en 2021 dans le domaine de la restauration de livres anciens. Ensemble, elles entendent comprendre de manière scientifique les raisons des dégradations de ce patrimoine presque quatre siècles après leur production. Le projet se déroulera en différentes étapes. « Ana dresse actuellement l’inventaire des exemplaires concernés et le répertoire de leurs caractéristiques matérielles et de leurs dégradations », continue Catherine Charles. « Ensuite, l’espèce animale du parchemin sera identifiée par une méthode de bioarchéologie non invasive ».
La recherche pourra compter en cela sur l’expertise développée à l’UNamur dans le cadre du projet Pergamenum21, piloté par le professeur Olivier Deparis (Département de physique, institut NISM), qui portait sur l’analyse des parchemins médiévaux de l’abbaye d’Orval.
« L’origine du parchemin sera déterminée avec l’aide de Marc Dieu, gestionnaire de la plateforme de spectrométrie de masse MaSUN de l'UNamur. On constate en effet que les parchemins des reliures rétrécissent avec le temps. Cela est-il accentué par l’origine animale, veau, chèvre, mouton ? ».
Les volumes prendront ensuite la direction du CHU CHU UCL Namur, où ils seront radiographiés – technique une fois encore non invasive – avec l’aide de Jean-François Nisolle, professeur à la Faculté de médecine de l’UNamur ( Institut PaTHs) et chef de clinique au CHU.
« On confrontera ensuite les données issues de l’étude visuelle de la structure de la reliure aux analyses bioarchéologiques et en imagerie afin de confirmer ou non nos hypothèses», conclut Catherine Charles.
Les résultats du projet pourront alors donner lieu à la mise en place de pratiques de restauration mieux adaptées à ce patrimoine. Les connaissances acquises dans le cadre du projet seront également exploitées pour améliorer les reliures dites « de conservation », en cernant de plus près les facteurs qui influencent à long terme une meilleure préservation des ouvrages anciens.

A prestigious publication for an international microbiology research team
A prestigious publication for an international microbiology research team
The team of professor Xavier De Bolle has just published an article in the prestigious EMBO Journal published by Springer Nature. His discovery? A lipid transport channel through the cell membrane of Brucella, the bacteria responsible for Brucellosis in cattle. This finding could be used to generate attenuated strains of the bacteria; a process used in vaccine manufacturing.

The study of Brucella is one of the areas of microbiology in which UNamur research teams have specialised for more than twenty years. This pathogenic bacteria infects livestock, causing Brucellosis, a disease that can be transmitted to humans through the consumption of unpasteurised dairy products, contact with infected animal tissue or inhalation. It is one of the most widespread zoonoses in the world.
A catalogue of weaknesses
Over the years of research, the team led by Professor Xavier De Bolle, Professor in the Department of Biology, member of the Microorganism Biology Research Unit (URBM) at the University of Namur and investigator at the WEL Research Institute, has been able to establish a kind of “catalogue” of Brucella's weaknesses.
There are millions of species of bacteria. It is therefore impossible to study them on a case-by-case basis. However, they have similar behaviours and functions, which is why a specific bacterium is considered the model: Escherichia coli (or E. coli), an intestinal bacteria found in warm-blooded organisms. Discovered in 1885, it has been the subject of numerous studies and has therefore become a reference in scientific literature.
A bacterium such as E. coli or Brucella is like an onion with only two layers (inner and outer membrane) and a central element (the cytoplasm) in which the organism produces everything it needs. It also exports elements to the outside, particularly lipids. However, these processes are still poorly understood, even for E. coli. Nevertheless, they represent an interesting avenue to explore to reach the heart of the pathogenic bacteria.
Mutants pave the way
During this research, which combined bacteriology and genetics, the researchers developed a technology that allows them to induce mutations in Brucella and also to identify where these mutations are located. They succeeded in creating a mutant that showed them the way to a veritable ‘lipid tunnel’, a passageway in the inner and outer membrane walls of the bacteria.

This provides valuable information about a new weakness in the pathogen and therefore provides information on how to combat it more effectively.

Our research, funded by the FNRS and the WEL Research Institute, is fundamental. Of course, we are still in the very early stages of developing applied processes. But we are taking another step towards unlocking the secrets of Brucella's armour so that one day we can hope to control the disease with an effective vaccine
An international research team
- Adélie Lannoy (currently post-doc in Toulouse) and Xavier De Bolle (URBM, UNamur – Belgium)
- Marc Dieu and Patsy Renard (UNamur, URBC - Belgium)
- Antonia García Fernández (Centro de Metabolómica y Bioanálisis (CEMBIO), Facultad de Farmacia, Universidad San Pablo-CEU – Spain)
- Raquel Condez-Alvarez (Department of Microbiology and Parasitology, Instituto de Investigación Sanitaria de Navarra (IdiSNA)University of Navarra – Spain)
The researchers would like to thank the FNRS and the WEL Research Institute for their support in this research. They would also like to thank the ‘Mass Spectrometry – MaSUN’ platform and the ‘Level 3 Biosafety Laboratory – BL3’ technology platform at UNamur for their cutting-edge equipment and expertise.
Xavier De Bolle - Mini CV
Xavier De Bolle presented his doctorate at UNamur (FUNDP at the time) in 1995. After 4 years of post-doc, including one at the University of Oxford, he joined URBM (Biology, Sciences), where he leads a research group. He has supervised 25 doctoral theses (including 4 in progress) and some of his former students have generated their own research team (at the UNamur, the UCLouvain, the VUB).
Professor Xavier De Bolle is a member of the URBM in the Department of Biology at the Faculty of Science of the University of Namur. He is a member of the Namur Research Institute for Life Sciences (Narilis).
Since June 2024, he is also a researcher at the WEL Research Institute for the Welbio-Health programme in the field of ‘Microbiology and Infectious Diseases’.
This work illustrates the cutting-edge expertise developed at UNamur in the field of molecular microbiology, an area in which UNamur has decided to invest by organising a master's degree in molecular microbiology since September 2019. This is a unique master's degree in Europe, taught entirely in English and rooted in research.
Find out more
- New clues to break through Brucella's armour (02/27/2023) - https://unamur.be/fr/newsroom/de-nouveaux-indices-pour-percer-larmure-de-brucella
- Alison Forrester and Xavier De Bolle awarded grants from the latest FRFS-Welbio call for projects (07/23/2024) - https://unamur.be/fr/newsroom/laureats-dernier-appel-projets-frfs-welbio

Parchment bindings under the microscope
Parchment bindings under the microscope
To restore an old book correctly, it is essential to know the secrets of its manufacture and the reasons for its deterioration. Thanks to the King Baudouin Foundation's Jean-Jacques Comhaire Fund, the restoration workshop of the Moretus Plantin University Library has launched a new research project on parchment bindings in the Southern Netherlands in the 16th and 17th centuries. The aim is to gain a better understanding in order to improve conservation.

La Bibliothèque Universitaire Moretus Plantin (BUMP) a la chance de bénéficier de son propre atelier de restauration, qui permet à la fois une conservation optimale des ouvrages et leur mise à disposition des scientifiques et du public dans de bonnes conditions. « En restauration, on doit généralement constater les détériorations sans connaître leurs origines. Pourtant, comprendre le mouvement séculaire d’une reliure permettrait d’adapter nos interventions afin qu’elles soient les plus adéquates vis-à-vis de cette évolution », explique Catherine Charles, restauratrice responsable de l’atelier de la BUMP.
Le nouveau projet financé par le Fonds Jean-Jacques Comhaire, qui soutient la recherche dans le domaine de l’archéométrie, vise à apporter des réponses aux questions que se posent les restaurateurs sur les reliures souples en parchemin réalisées dans nos régions aux Temps Modernes. « La BUMP conserve un corpus représentatif de ces reliures, et en particulier de reliures couvrant des volumes qui ont en commun le fait d’avoir été imprimés à Anvers, chez Plantin-Moretus, aux 16e et 17e siècles », indique Catherine Charles. « Dans cet ensemble cohérent de plus de 200 livres, nombreux sont les cas où une dégradation similaire est constatée : une courbure du dos de plus en plus accusée, ce qui fend les mors et fait ressortir le corps d’ouvrage. Le projet de recherche est l’opportunité de comprendre la construction matérielle de ces livres et de faire le lien ou non avec leur dégradation progressive ».
Le Fonds Comhaire rend possible l’engagement, aux côtés de Catherine Charles, d’Ana Oñate Muños, assistante de recherche diplômée de La Cambre en 2021 dans le domaine de la restauration de livres anciens. Ensemble, elles entendent comprendre de manière scientifique les raisons des dégradations de ce patrimoine presque quatre siècles après leur production. Le projet se déroulera en différentes étapes. « Ana dresse actuellement l’inventaire des exemplaires concernés et le répertoire de leurs caractéristiques matérielles et de leurs dégradations », continue Catherine Charles. « Ensuite, l’espèce animale du parchemin sera identifiée par une méthode de bioarchéologie non invasive ».
La recherche pourra compter en cela sur l’expertise développée à l’UNamur dans le cadre du projet Pergamenum21, piloté par le professeur Olivier Deparis (Département de physique, institut NISM), qui portait sur l’analyse des parchemins médiévaux de l’abbaye d’Orval.
« L’origine du parchemin sera déterminée avec l’aide de Marc Dieu, gestionnaire de la plateforme de spectrométrie de masse MaSUN de l'UNamur. On constate en effet que les parchemins des reliures rétrécissent avec le temps. Cela est-il accentué par l’origine animale, veau, chèvre, mouton ? ».
Les volumes prendront ensuite la direction du CHU CHU UCL Namur, où ils seront radiographiés – technique une fois encore non invasive – avec l’aide de Jean-François Nisolle, professeur à la Faculté de médecine de l’UNamur ( Institut PaTHs) et chef de clinique au CHU.
« On confrontera ensuite les données issues de l’étude visuelle de la structure de la reliure aux analyses bioarchéologiques et en imagerie afin de confirmer ou non nos hypothèses», conclut Catherine Charles.
Les résultats du projet pourront alors donner lieu à la mise en place de pratiques de restauration mieux adaptées à ce patrimoine. Les connaissances acquises dans le cadre du projet seront également exploitées pour améliorer les reliures dites « de conservation », en cernant de plus près les facteurs qui influencent à long terme une meilleure préservation des ouvrages anciens.



